Forest plots
Overview
Teaching: 0 min
Exercises: 0 minQuestions
FIXME
Objectives
FIXME
hvad er de?
Forest plots (der ikke har noget med skove at gøre) er plots der viser de estimerede resultater fra flere videnskabelige studier - af samme fænomen. Sammen med de samlede resultater. De bruges i metaanalyser af litteraturen. Og kaldes også blobbogrammer.
Hvordan laver man dem?
Man starter med at lade den rigtige funktion beregne odds ratioer for ens studier.
annoteret
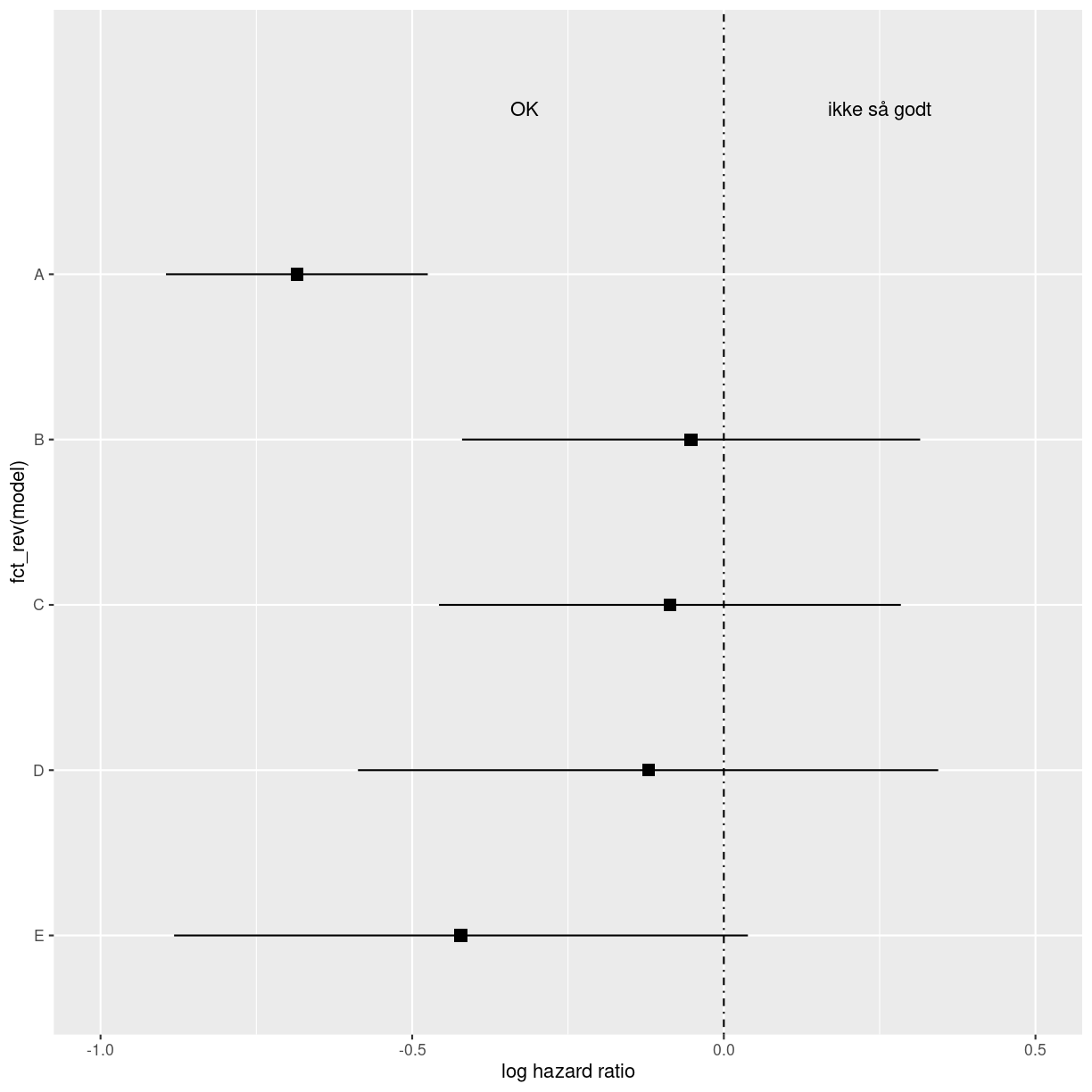
vi vil godt have p-værdier på dem. så vi starter med at få trukket data ud på de forskellige modeller. Her er noget data vi har forberedt på forhånd. Der er fem modeller, der er estimater, lav og høj ende af konfidensintervallerne, p-værdier. Og (den naturlige) logaritme af estimat og konfidensintervaller:
data <- tribble(
~model, ~log.estimate, ~log.conf.low, ~log.conf.high, ~estimate, ~conf.low, ~conf.high, ~p.value,
"A", -0.685, -0.895 , -0.475 , 0.504 , 0.409 , 0.622, 1.68e-10,
"B", -0.0526, -0.420 , 0.315 , 0.949 , 0.657 , 1.37, 7.79e-1,
"C", -0.0864 , -0.457 , 0.284 , 0.917 , 0.633 , 1.33, 6.47e-1,
"D", -0.121 , -0.587 , 0.344 , 0.886 , 0.556 , 1.41, 6.09e-1,
"E", -0.422 , -0.882 , 0.0386 , 0.656 , 0.414 , 1.04, 7.26e-2
)
forestplot(base_data)
Error in eval(expr, envir, enclos): object 'base_data' not found
Det skal vi have plottet:
midt <- data %>%
ggplot(aes(y = fct_rev(model))) +
# theme_classic() +
geom_point(aes(x =log.estimate), shape = 15, size = 3) +
geom_linerange(aes(xmin = log.conf.low, xmax = log.conf.high)) +
labs(x = "log hazard ratio") +
coord_cartesian(ylim=c(1,6), xlim=c(-1,.5)) +
geom_vline(xintercept = 0, linetype = "dotdash") +
annotate("text", x = -.32, y = 6, label = "OK") +
annotate("text", x = 0.25, y = 6, label = "ikke så godt")
midt
plot of chunk unnamed-chunk-3
Det er det plot vi vil have i midten, så det gemmer vi.
Så har vi noget vi vil se til venstre for det plot:
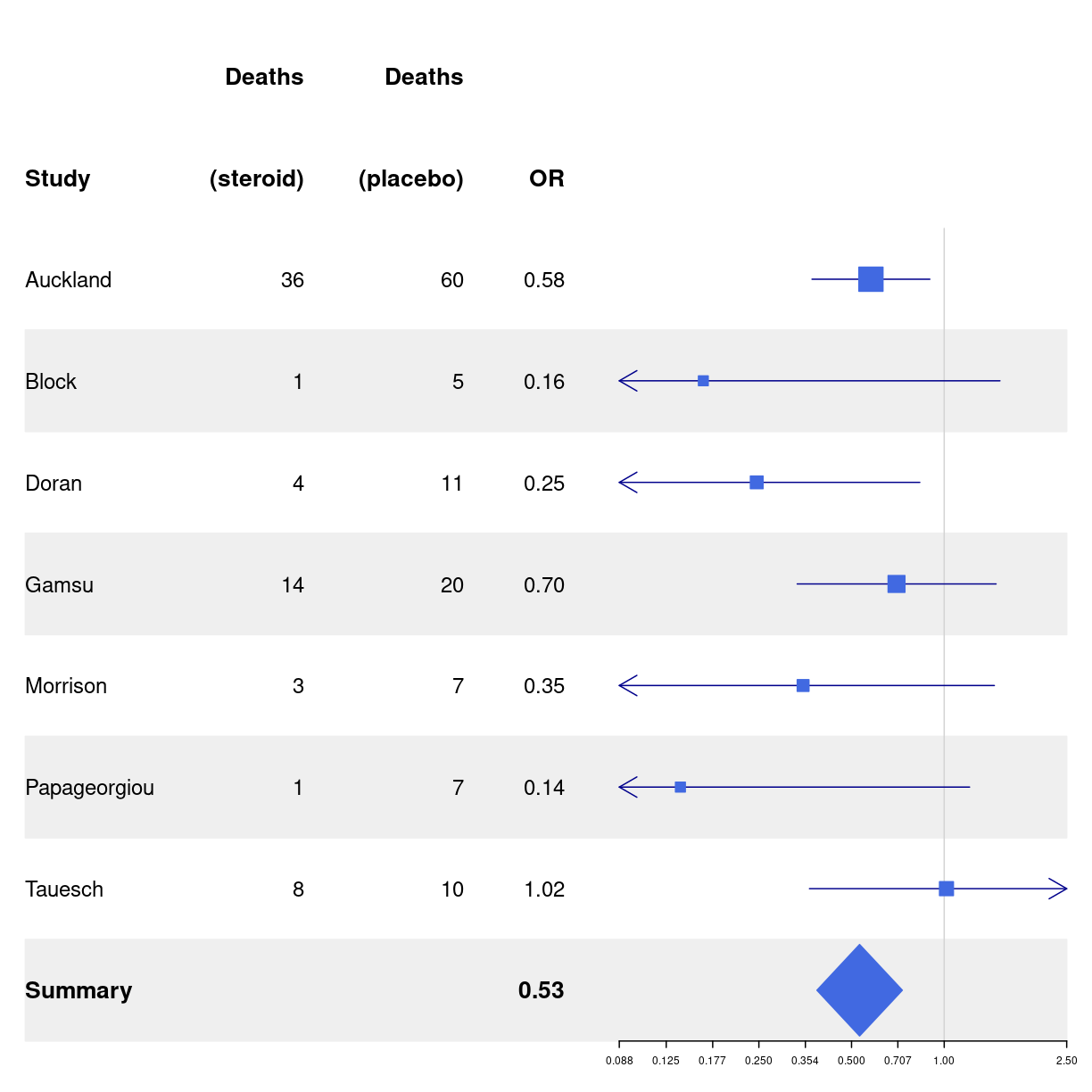
# Cochrane data from the 'rmeta'-package
base_data <- tibble::tibble(mean = c(0.578, 0.165, 0.246, 0.700, 0.348, 0.139, 1.017),
lower = c(0.372, 0.018, 0.072, 0.333, 0.083, 0.016, 0.365),
upper = c(0.898, 1.517, 0.833, 1.474, 1.455, 1.209, 2.831),
study = c("Auckland", "Block", "Doran", "Gamsu",
"Morrison", "Papageorgiou", "Tauesch"),
deaths_steroid = c("36", "1", "4", "14", "3", "1", "8"),
deaths_placebo = c("60", "5", "11", "20", "7", "7", "10"),
OR = c("0.58", "0.16", "0.25", "0.70", "0.35", "0.14", "1.02"))
log(36/60)
[1] -0.5108256
base_data %>%
forestplot(labeltext = c(study, deaths_steroid, deaths_placebo, OR),
clip = c(0.1, 2.5),
xlog = TRUE) |>
fp_set_style(box = "royalblue",
line = "darkblue",
summary = "royalblue") %>%
fp_add_header(study = c("", "Study"),
deaths_steroid = c("Deaths", "(steroid)"),
deaths_placebo = c("Deaths", "(placebo)"),
OR = c("", "OR")) |>
fp_append_row(mean = 0.531,
lower = 0.386,
upper = 0.731,
study = "Summary",
OR = "0.53",
is.summary = TRUE) %>%
fp_set_zebra_style("#EFEFEF")
plot of chunk unnamed-chunk-5
Key Points
FIXME